超過死亡2: 感染研が採用しているFarringtonアルゴリズムの利用方法
正しい超過死亡の算出の仕方は?
- 超過死亡の計算手法いくつもあり、これが決定打というものは無い
- 感染研はFarringtonアルゴリズム[1]を採用
- アルゴリズムが何であれ、ワクチンの影響を除外して論じるのなら不適切
- どの年を基準に予測値を出し何を検出しようとするかが問題
- 図は感染研超過死亡の推定Q&A[2]と超過死亡の推定(データ分析)[3]より

ここでアルゴリズム自体が不適切と指摘をするつもりは無い。しかし適用方法により不適切になるということは指摘したい。現に超過死亡はワクチンは関係無いという前提で利用されていることがその証左でもある。
何を検出しようとするかに加えて今後はどの年を基準に予測値を出すかが問題になっていくと筆者は考えている。ワクチンの影響を調べようというのに、ワクチンを射って影響が出ていると思われる年を含めて比較対照にするのでは影響が隠されてしまう。
どの年を基準にするかで予測値が変るという話は次回以降にして、本記事は、興味のある方もいると思うので、Farringtonアルゴリズムの利用方法について筆者が把握していることにつて説明する。純粋にアルゴリズムの概要や利用方法を説明するのが目的であり、オリジナルのコードが改変されていることも含めて、批判する意図は一切無い。
解説/Explanation
超過死亡の計算手法いくつもあり、国立感染症研究所(以下、感染研)も『ゴールドスタンダードと呼べるものは無い』[2]としている。その中で感染研はCDCも採用しているFarringtonアルゴリズム[1]を採用している。過去の数年間の同じ週や月の死亡数から、ある週や月の死亡数を予測し、その差を超過死亡数としている。もう少し詳しく説明すると、95%予測区間と幅を持たせて予測しその幅を超えたときを超過死亡とする。ただし予測区間では無く予測値を超えたときの数値も拾うような使い方になっている。詳しくは鈴村氏のアゴラ記事「超過死亡と接種後死亡について再び考察(前編)」[4]を参照されたい。
感染研はFarringtonアルゴリズムをRコードにより利用
- アルゴリズムはFarringtonの1996年の論文[1]が基
- 感染研が公開しているページ[3]に計算のためのRコードが記載[5]
- surveilance パッケージを読込み farringtonFlexible 関数を呼出し
- このRコード自体の中では複雑な計算は何もしていない
- 図は感染研提供のRコード(ただし旧版)

解説/Explanation
アルゴリズムはFarringtonの1996年の論文[1]が基になっている。筆者はよく誤解されるが統計の専門家ではないため、アルゴリズムの詳細については理解できていない。しかし情報処理技術者ではあるので利用することはできる。
Rプロジェクトの一貫としてFarringtonアルゴリズムが提供
- RプロジェクトはSurveillanceパッケージを提供
- パッケージ中にFarringtonアルゴリズムの実装farringtonFlexible関数が含まれる
- 感染研はこのパッケージを利用して超過死亡を計算
- 図はSurveillanceパッケージのページ[6]とFarringtonアルゴリズム詳細説明[7]
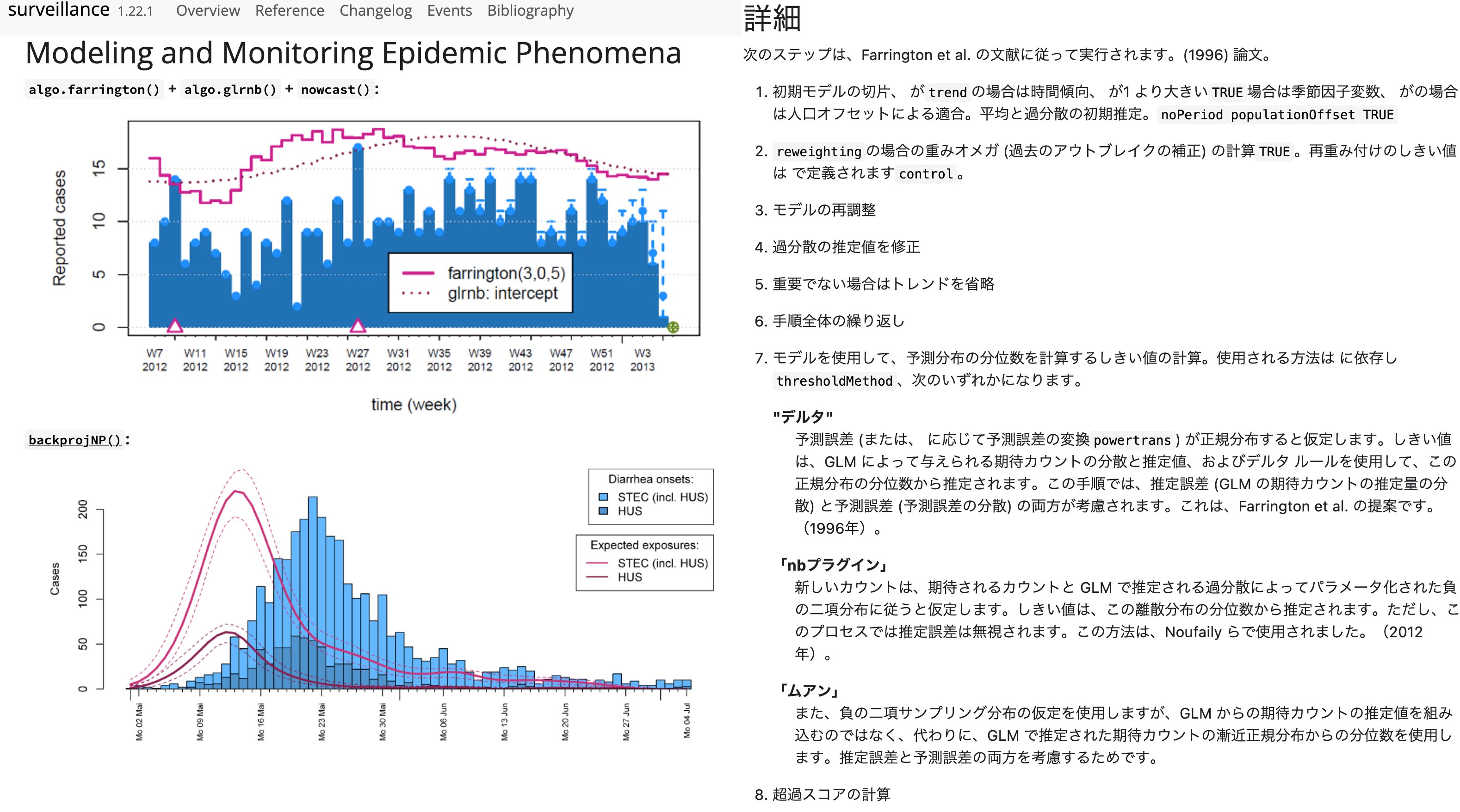
感染研はここでメンテナンスされているパッケージを利用して超過死亡を計算しているというわけである。
解説/Explanation
既に説明したように、感染研が利用しているFarringtonアルゴリズムの実装であるfarringtonFlexible関数はRプロジェクトのSurveillanceパッケージ[6]で提供されている。図はそのトップページとFarrigntonアルゴリズムの説明[7]の日本語訳である。コードが読める方は論文[1]を読む前にこの説明と実際のコードを見た方が理解が早いと思う。
感染研のRコードでグラフを描いてみる
- 超過死亡ダッシュボード[8]で提供の最新CSVを用いグラフを描く
- Rコード[5]が古いためかフィールド名に差異あるので修正
- 全都道府県と全国の死者数と超過死亡を示すグラフが生成される
- 図は感染研のRコードで描いた北海道のグラフ(ただし年月間違い)
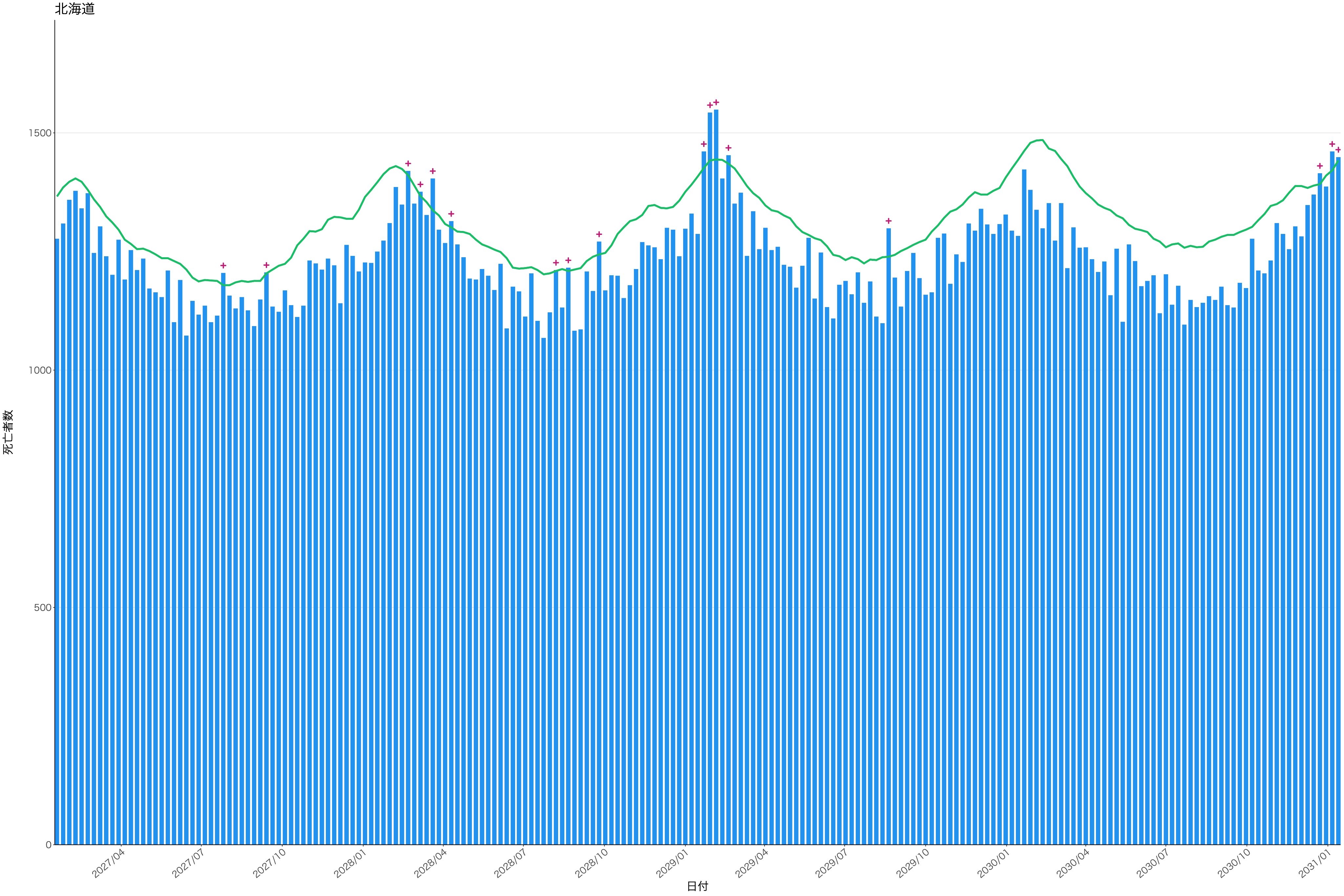
上に示したのは少し古いコードだったためか、超過死亡ダッシュボード[8]で公開されている感染研の最新CSVデータファイルはフィールド名が変っている箇所がありその儘では使えない。CSVデータ中の prefecture_EN を prefectureEN に、Obvserved を count に変更すれば動かすことができた。
なおRコードの置いてあるページ[2]にもCSVデータファイルがあるのだがこちらもRコード[5]の想定しているフィールド名と一致していなかった。
図のグラフはRコードを起動した結果表示された北海道の死亡数と超過死亡を示すグラフである。この他、全都道府県と全国のグラフが生成される。ただ修正が足りていないためか年月がおかしくなっている。しかし動作確認が目的なのでこれ以上は追求しない。
解説/Explanation
感染研のRコードでグラフを描いてみる。
超過死亡ダッシュボードのRコード
- 最新と思われるRコード[9]は古いコードに比べて随分長い
- farringtonFlexible関数を全てコピーし必要な箇所だけ修正
- 本質的な変更は無いが最新のコードを最初に見てしまうと全体像把握が困難
- 図はGitHub上にある元のコード[10]
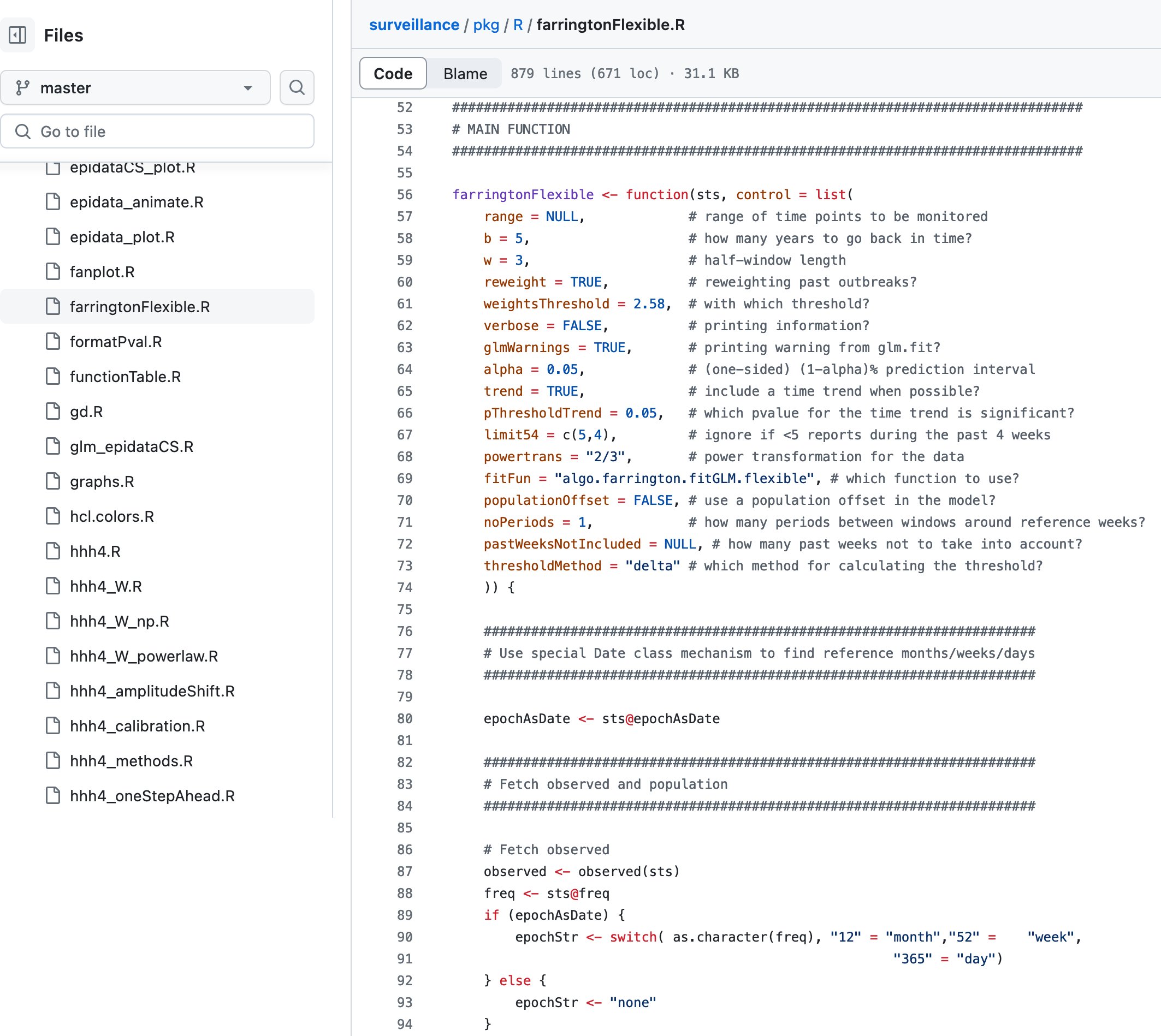
超過死亡ダッシュボード[8]で利用されている最新と思われるRコード[9]はダッシュボードのトップページに添付されている。古いコードに比べて随分長くなっているが、本質的な部分が長くなっているわけでは無い。元はパッケージに含まれていたfarringtonFlexible関数に独自に修正を加えるため、関連関数を全てコピーし、必要な箇所だけ修正しているためである。(コードのコピーと改変自体は何ら批難されるものでは無いが、公開する以上、元のソースコードとGPLv2ライセンスに従うことを明示すべきだと思う)
また最新と思われるRコードは以前と違いグラフ作成は行わず計算結果をデータとして保持するだけで、書き出しは別途行う必用がある。この変更は以前はグラフの画像をWEBに貼る運用だったが、ダッシュボードのWEBインタフェースから様々なグラフを動的に提供するように変更したことへの対応によるものだろう。
以上、筆者が把握している感染研でのFarringtonアルゴリズム利用方法の説明である。
解説/Explanation
実際、farrigtonFlex関数やそこから呼出される関数は、図に示したGitHub上にある元のコード[10]とほとんど同じである。感染研の最新のコードを最初に見てしまうと全体像が把握しにくくなると判断したので、旧版のRコード[5]から解説したというわけである。
参考文献
- FARRINGTON, C. P., et al. A statistical algorithm for the early detection of outbreaks of infectious disease. Journal of the Royal Statistical Society: Series A (Statistics in Society), 1996, 159.3: 547-563., https://rss.onlinelibrary.wiley.com/doi/abs/10.2307/2983331
- 国立感染症研究所, 超過死亡の推定に関するQ&A(2020年8月31日時点版), https://www.niid.go.jp/niid/ja/from-idsc/493-guidelines/9834-excess-mortality-qa-200831.html
- 我が国における超過死亡の推定(2020年4月までのデータ分析), https://www.niid.go.jp/niid/ja/from-idsc/493-guidelines/9748-excess-mortality-20jul.html
- 鈴村 泰, アゴラ記事, 超過死亡と接種後死亡について再び考察(前編), 2021/12/21, https://agora-web.jp/archives/2054382.html
- 国立感染症研究所, 超過死亡の推定に関するQ&A(2020年8月31日時点版)補足資料1:FarringtonアルゴリズムのRコード, https://www.niid.go.jp/niid/images/epi/excess_mortality/results200731/suppl1_R.txt
- The R Project for Statistical Computing, Surveillance, https://surveillance.r-forge.r-project.org/pkgdown/index.html
- The R Project for Statistical Computing, Surveillance for Univariate Count Time Series Using an Improved Farrington Method, https://surveillance.r-forge.r-project.org/pkgdown/reference/farringtonFlexible.html
- 日本の超過および過少死亡数ダッシュボード, https://exdeaths-japan.org/graph/weekly
- 日本の超過および過少死亡数ダッシュボード, 補足資料 R_code.R, https://exdeaths-japan.org/data/R_code.R
- GitHub, r-forge/surveillance/pkg/R/farringtonFlexible.R, https://github.com/r-forge/surveillance/blob/master/pkg/R/farringtonFlexible.R
- 国立感染症研究所, 超過死亡の推定に関するQ&A(2020年8月31日時点版), https://www.niid.go.jp/niid/ja/from-idsc/493-guidelines/9834-excess-mortality-qa-200831.html