Speicher氏によるDNA汚染発見と有害事象との関連 (2023/10/09 WCH DNA汚染問題緊急専門者会議)
27本ものmRNAワクチンを調べ全てからDNA汚染発見
- Speicher氏はカナダのゲルフ大学病理生物学学部所属、共著McKernan氏
- 蛍光光度計 Qubit(横軸) では 1,896〜5,100ng/dose で規制値 10ng/dose の 188倍から509倍上回る
- qPCR(縦軸)では規制値以下(spikeプライマー 0.22 - 2.43 ng/dose)
- グラフはプレプリントSpeicher2024[2]より
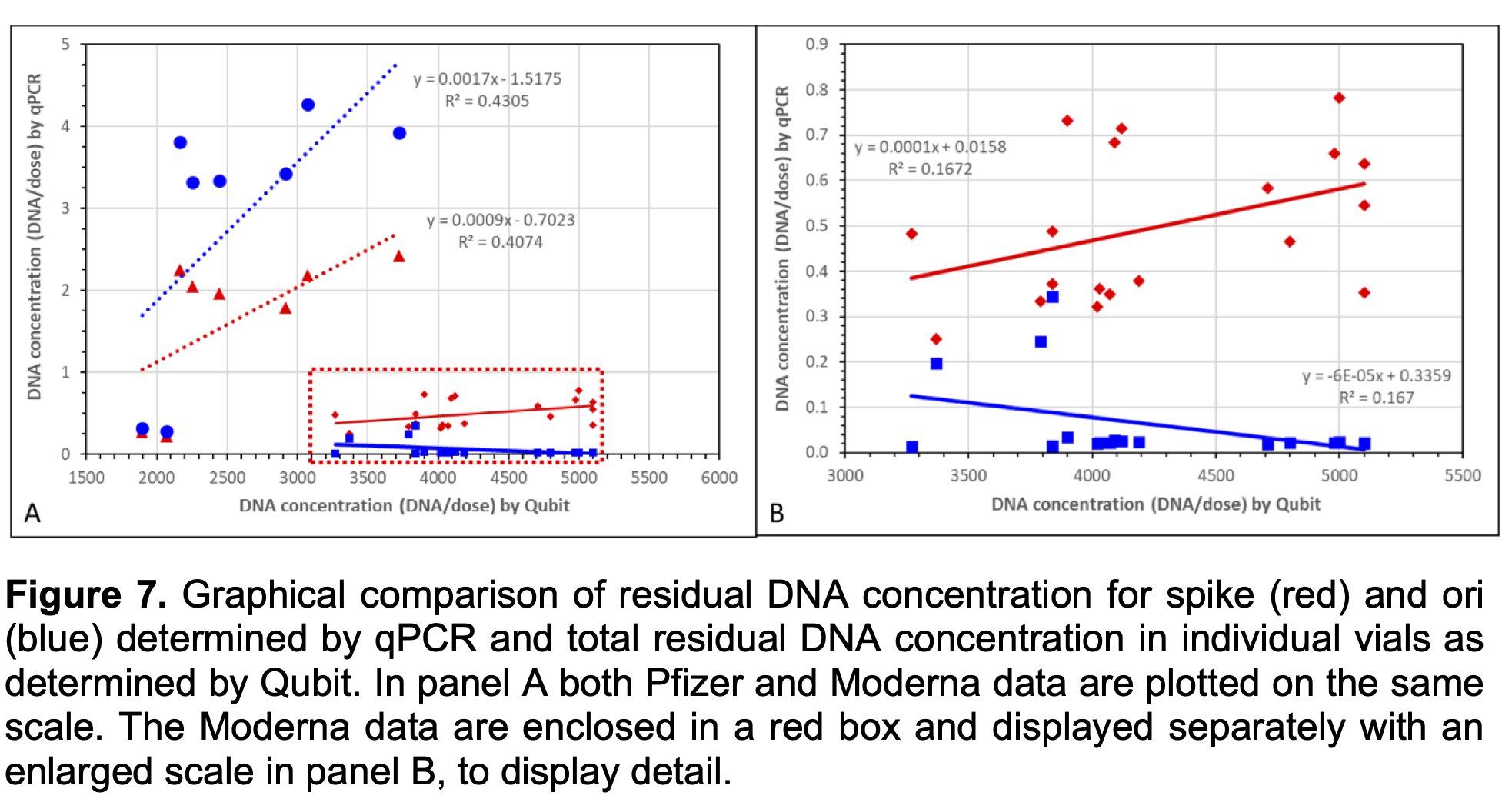
解説/Explanation
結果と結論の翻訳『結果:プラスミド起源の複製(ori)およびスパイク配列の定量周期(Cq)値(1:10 希釈)は、Pfizer では 18.44 - 24.87 および 18.03 - 23.83、Moderna では 22.52 – 24.53 および 25.24 – 30.10 でした。これらの値は、ori および spike について、それぞれ qPCR によって測定された 0.28 – 4.27 ng/dose および 0.22 - 2.43 ng/dose(Pfizer)、0.01 - 0.34 ng/dose および 0.25 – 0.78 ng/dose(Moderna)に対応しています。さらに、Pfizer および Moderna について、Qubit® フルオロメトリーによって測定された値は 1,896 – 3,720 ng/dose および 3,270 – 5,100 ng/dose でした。SV40 プロモーター強化 ori は Cq スコアが 16.64 – 22.59 の Pfizer 試験管でのみ検出されました。探索的分析において、投与あたりの DNA 量と重大な有害事象(SAE)の頻度との間に投与応答関係の初期証拠が見つかりました。この関係は Pfizer 製品と Moderna 製品で異なっていました。サイズ分布分析では、平均 DNA フラグメント長が 214 塩基対(bp)で、最大は 3.5 kb でした。プラスミド DNA はおそらく LNPs 内に存在し、核酸酵素から保護されています。
結論:これらのデータは、これらのワクチンの各投与あたりの DNA 分子が数十億から数千億存在することを示しています。フルオロメトリーを使用すると、すべてのワクチンが、FDA および WHO によって設定された 10 ng/dose の残存 DNA に関するガイドラインを 188倍から 509倍も上回っています。ただし、すべてのワクチンの qPCR 残存 DNA 含有量はこれらのガイドラインを下回っており、定量的なガイドラインを解釈する際の方法論的な明確さと一貫性の重要性を強調しています。qPCR で測定された残存 DNA と SAE の投与応答効果の初期証拠は確認とさらなる調査が必要であるということを示唆しています。私たちの調査結果はワクチンの安全性に関する既存の懸念を拡大し、LNPs を使用した効率的な DNA 転写が導入される前に考案されたガイドラインの関連性に疑問を投げかけています。いくつかの明白な制約があるにもかかわらず、我々は私たちの研究が法的な条件の下で複製され、効率的な DNA 転写と累積的な投与量を考慮したガイドラインの改訂を要望しています。』
蛍光光度計 Qubit(横軸) では 1,896〜5,100ng/dose で規制値 10ng/dose の 188倍から509倍上回る DNA汚染が検出された。
qPCR(縦軸)では全てが規制値 10ng/dose 以下となった。spikeプライマー(赤)での結果は 0.22 - 2.43 ng/dose であった。spikeプライマーとoriプライマーとで結果が随分と違うようであった。
解説/Explanation
David J. Speicher氏は、カナダのゲルフ大学の病理生物学学部所属でありmRNAワクチン27本も調べその全てからDNA汚染を発見したというプレプリントを公開[2]した。共著にはMcKernan氏もいる。2023/10/09 に開催された WCH DNA汚染問題緊急専門者会議において、[発表自体は McKernan氏]により行われた。](Tonakai202310101)
qPCRの結果と有害事象との関連
- グラフの上部分はqPCRの結果によるDNA汚染量で赤がSpike青がORI
- 全て基準値以下となっているがqPCRなので過少評価
- 下部分は各ロットごとの有害事象
- DNA汚染量が多いと有害事象が多くなってるのではと関連性が疑われる
- グラフはプレプリントSpeicher2024[2]より
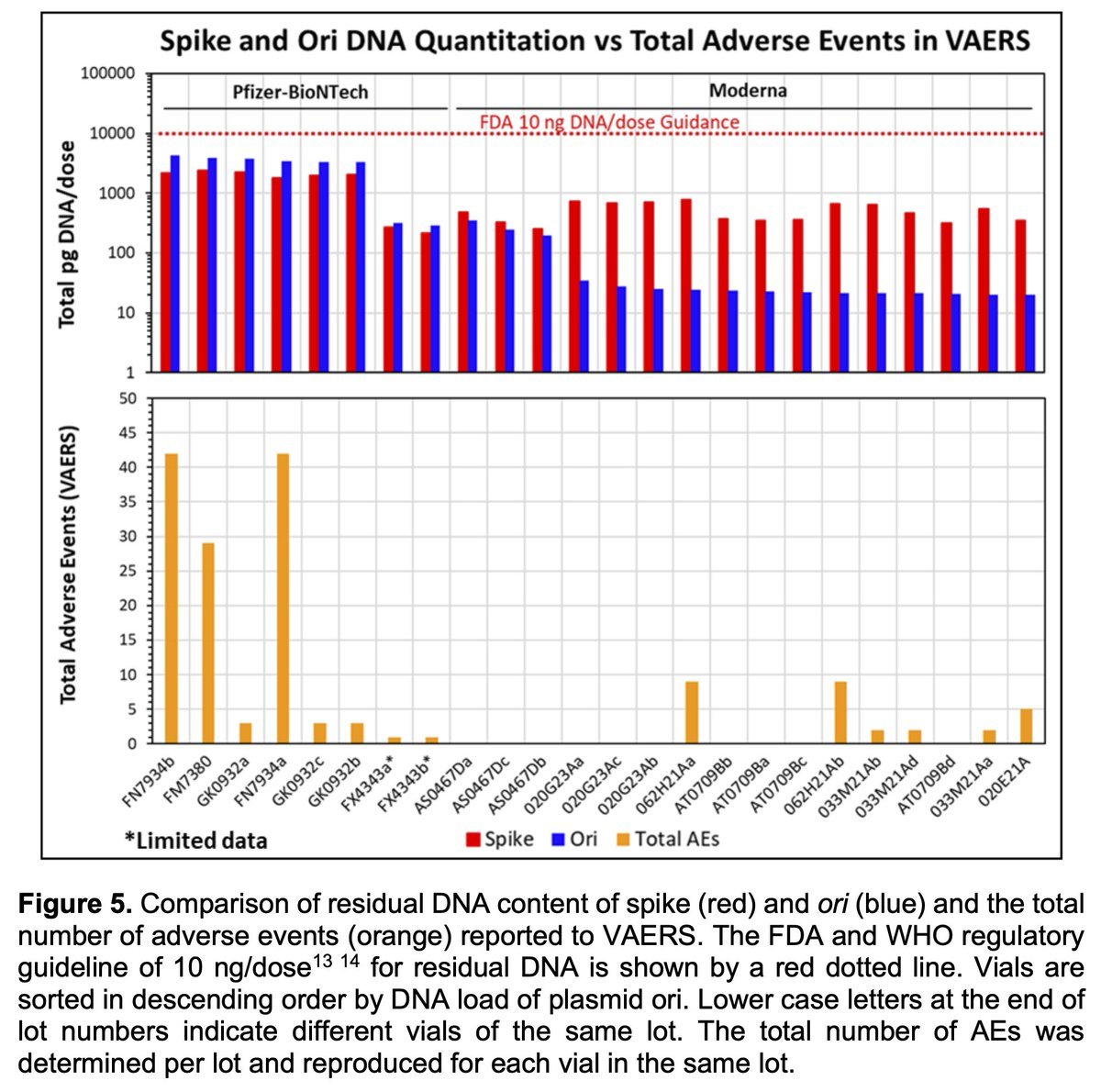
下部分はDNA汚染を検出したロットごとの有害事象件数であり、DNA汚染量が多いと有害事象が多くなってるのではと関連性が疑われるグフラフとなっている。
解説/Explanation
グラフの上部分はロットごとのqPCRの結果によるDNA汚染量であり、赤がSpikeプライマーで青がORIプライマーの結果を示している。全て基準値以下となっているが、これはqPCRでの検出結果であり過少評価となっている。
おまけ: RNAが大量に存在する場合にQubitでDNA量をを測定できるか?
- RNAだけでもDNAと7%は誤認されるというメーカーのグラフの二つの解釈
- 1. メーカーがDNA蛍光染料がいくら特異的といっても対RNAで7%誤検出
- 2. テストに使われたRNAの純度が甘く7%ほどDNAが含まれている
- どちらにせよMcKernan氏結果DNA/RNA比2%〜12%、Qubitで計測は可能
- 誤検出最大値は2%で、差し引いても最大で対RNA比10%のDNAが検出
- Qubitは世界中でDNA測定に使われており、RNAが多いから使えないと早々に結論づけるのは、科学者のやることでは無い
- グラフはQubit販売メーカーページ[3]からのグラフに追記
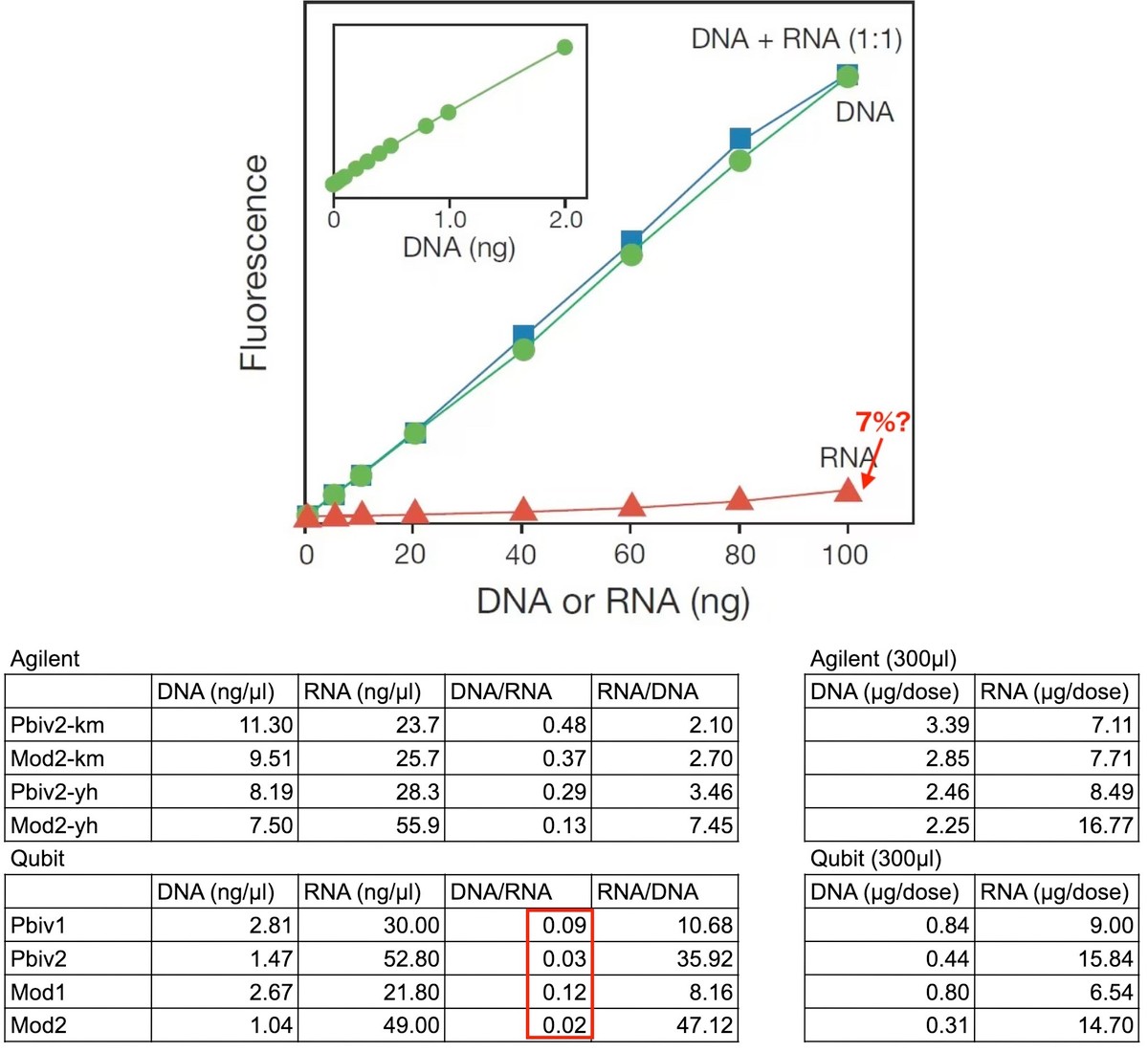
グラフは、蛍光染料を使うQubitだとRNAだけでを検量してもDNAだと7%は誤認されると解釈されるメーカーのグラフである。
しかし解釈はにはもう一つある。それは、テストに使われたRNAの純度が甘く7%ほどDNAが含まれている、というものである。
真偽を確かめるべく、実際にグラフを作成したメーカーに問い合せした。そして次の回答を得ている『質問いただいたdsDNA assay kitにおけるRNAの影響について、実験に利用したRNAは精製後にDNase処理を行ったものにはなるとのことです。ただDNAの残存が全くないかを確認することは難しく、残存が全くないか?という点については否定することができません』
つまりRNAだけの試薬を作るさいにDNAを完全に除去すること自体が難しいのであり、グラフはRNAをDNAと誤検出したのでは無く、混入してしまったDNAを正しく検出したのかもしれないのである。
どちらにせよ、McKernan氏の結果がDNA/RNA比 2%〜12% であるので、Qubitで計測は可能と考えている。つまり本環境では非特異性、RNAがDNAと検出される最大値は 2% と考えている。その最大値を引いても最大で対RNA比 10% のDNAが検出されたと見做される。
筆者の考察は間違ってるいるかもしれないが、間違っているにせよ、Qubitは世界中でDNA測定に使われており、RNAが多いから使えないと早々に結論づけるのは科学者のやることでは無い。工夫して検出する方法を考えるのが真の科学者の姿である。
解説/Explanation
SNS上ではQubitでRNAが大量に含まれている場合にDNAは計測できないと吹聴し、DNA汚染の矮小化を図るグループが存在する。彼らの主張の何がおかしいかを解説する。なお筆者は彼らに直接、彼らの解釈がおかしいことを指摘しているが、筆者に対する回答から無い。
参考文献
- tonakai, ツイート, Part2 ケビン・マッカーナン氏, 2023-10-10, https://x.com/tonakai79780674/status/1711705599912931335
- SPEICHER, David J., et al. DNA fragments detected in monovalent and bivalent Pfizer/BioNTech and Moderna modRNA COVID-19 vaccines from Ontario, Canada: Exploratory dose response relationship with serious adverse events. 2023., https://osf.io/preprints/osf/mjc97
- Qubit dsDNA Quantification Assay Kits, https://www.thermofisher.com/order/catalog/product/jp/ja/Q32851
- SPEICHER, David J., et al. DNA fragments detected in monovalent and bivalent Pfizer/BioNTech and Moderna modRNA COVID-19 vaccines from Ontario, Canada: Exploratory dose response relationship with serious adverse events. 2023., https://osf.io/preprints/osf/mjc97